DOI: http://dx.doi.org/10.19137/cienvet202224101
![]() Esta obra se publica bajo licencia Creative Commons 4.0 Internacional. (Atribución-No Comercial-
Compartir Igual) a menos que se indique lo contrario, http://www.creativecommons.org.ar/licencias.htm
Esta obra se publica bajo licencia Creative Commons 4.0 Internacional. (Atribución-No Comercial-
Compartir Igual) a menos que se indique lo contrario, http://www.creativecommons.org.ar/licencias.htm
ARTICULO DE INVESTIGACION
Estudio de asociación del polimorfismo del
gen de clase II BoLA-DQA1 con resistencia/
susceptibilidad a mastitis en ganado Holstein de
la provincia de La Pampa
Association study of the polymorphism of the BoLA
DQA1 class II gene with resistence/susceptibility
to mastitis in Holstein cattle from the province of
La Pampa
Estudo do polimorfismo do gene BoLA-DQA1
classe II e sua associação com resistência /
suscetibilidade à mastite em bovinos Holstein da
província de La Pampa
Baltian L1, Ripoli MV 2, Giovambattista G3
1 Facultad de Ciencias Veterinarias, Universidad Nacional de La Pampa, calle 5 esquina 116,
General Pico (6360), La Pampa.
2 Facultad Ciencias Naturales y Museo, Universidad Nacional de La Plata, Avenida 122 y 60,
La Plata (1900), Buenos Aires.
3 Facultad de Ciencias Veterinarias, Universidad Nacional de La Plata, calle 60 y 118 S/N, La
Plata (1900), Buenos Aires.
Correo electrónico: lbaltian@vet.unlpam.edu.ar
Resumen: La mastitis es una enfermedad frecuente en el ganado lechero que
conduce a una disminución en la producción y a un incremento en los
costos sanitarios. Esta enfermedad puede evaluarse a través del número de glóbulos blancos por mililitro de leche, conocido como conteo de células
somáticas (CCS). La variabilidad alélica de los genes del Complejo
Principal de Histocompatibilidad Bovino (Bovine Leukocyte Antigen,
BoLA) se ha asociado con resistencia y susceptibilidad a enfermedades
infecciosas. El objetivo del presente trabajo fue estudiar el polimorfismo
del gen de clase II BoLA-DQA1 y su asociación con resistencia /susceptibilidad
a mastitis medida a través del CCS en leche de ganado Holstein
de la provincia de La Pampa. La población se dividió en: 1) grupo caso,
con alto CCS y presencia de mastitis (≥ 250.000 cel/ml, susceptible) y
2) grupo control, con bajo CCS (< 250.000 cel/ml, resistente). El BoLADQA1
se genotipo en 60 animales mediante PCR-RFLP y PCR-SBT. El test
exacto de Fisher y Odds Ratio (OR) de Woolf-Haldane se utilizaron para
estudiar la asociación entre el CCS y las variantes alélicas. Se detectaron
16 alelos del gen BoLA-DQA1 y el alelo BoLA-DQA1*0101 evidenció un
OR de 4 (p = 0,058). Se destaca la importancia de los alelos del BoLADQA1
para seleccionar animales resistentes a enfermedades.
Palabras claves: BoLA-DQA1, resistencia, susceptibilidad, mastitis,
células somáticas
Abstract: Mastitis is a frecuently disease of dairy cattle that leads to a decrease
in production and an increase in health costs. This disease can be evaluated
through the number of white blood cells per milliliter of milk,
which is known as somatic cell count (CCS). Alleles of Bovine Leukocyte
Antigen (BoLA) genes were associated with resistance and susceptibility
to infectious diseases. The objective of this work was to study the
polymorphism of the class II molecules of BoLA-DQA1 and their association
with resistance/susceptibility to mastitis measured through the
CCS in milk in Holstein cattle from the province of La Pampa. The population
was divided into: 1) case group with high CCS and presence
of mastitis (≥ 250,000 cel/ml, susceptible) and 2) control group with
low CCS (<250,000 cel/ml, resistant). BoLA-DQA1 polymorphisms were
genotyped in sixty animals by PCR-RFLP and PCR-SBT. Woolf-Haldane’s
Fisher and Odds Ratio (OR) exact test were used to study the association
between CCS and allelic variants. 16 alleles of the BoLA-DQA1gene were
detected and the BoLA-DQA1*0101 allele showed an OR of 4 (p = 0.58).
This finding highlights the importance of BoLA-DQA1 alleles for selection
of resistant animals to infectious diseases.
Key words: BoLA-DQA1, resistance, susceptibility, mastitis, somatic cell
Resumo: A mastite é uma doença do gado leiteiro que leva à diminuição da
produção e a um aumento dos custos de saúde. Essa doença pode ser
avaliada por meio do número de glóbulos brancos por mililitro de leite,
conhecido como contagem de células somáticas (CCS). A variabilidade
alélica dos genes do Complexo Principal de Histocompatibilidade
Bovino (Bovine Leukocyte Antigen, BoLA) tem sido associada à resistência
e suscetibilidade a doenças infecciosas. O objetivo deste trabalho
foi estudar o polimorfismo do gene BoLA-DQA1 classe II e sua
associação com a resistência / suscetibilidade à mastite medida por
CCS em leite de gado Holstein da província de La Pampa. A população
foi dividida em: 1) grupo caso com CCS alto e presença de mastite (≥ 250.000 células / ml, suscetível) e 2) grupo controle com CCS baixo
(<250.000 células / ml, resistente). Foi determinado o genótipo do
BoLA-DQA1 em 60 animais por PCR-RFLP e PCR-SBT. O teste exato de
Fisher e o Odds Ratio (OR) de Woolf-Haldane foram usados para estudar
a associação entre CCS e variantes alélicas. Foram detectados 16
alelos do gene BoLA-DQA1 e o alelo BoLA-DQA1 * 0101 apresentou OR
de 4 (p = 0,058). Destaca-se a importância dos alelos BoLA-DQA1 para
selecionar animais resistentes a doenças.
Palavras-chave: BoLA-DQA1, resistência, suscetibilidade, mastite, células somáticas.
La mastitis es una reacción inflamatoria de la glándula mamaria
frecuente en el ganado lechero que daña el tejido secretor de leche.
Cuando la ubre se lesiona ocurre una inflamación con acumulación de
glóbulos blancos que, junto a células epiteliales del tejido productor
de leche, constituyen las células somáticas (CCS).
Por otra parte, la mastitis subclínica se caracteriza por la presencia
de microorganismos y un conteo elevado de células somáticas en la
leche, pudiendo no tener tratamiento (1) y sin alteraciones evidentes
en la ubre (2). Pero sí ocurren cambios en la composición de la leche a
causa de las bacterias y componentes inflamatorios (3).
El número de células somáticas puede ser usado como un índice
para evaluar mastitis ya que existe una alta correlación entre un número
elevado de células somáticas y presencia de mastitis (4–6). Además,
junto a los recuentos bacterianos en tanques a granel, los residuos de antibióticos y los valores nutricionales son mediciones de la calidad
de la leche (7).
Los cambios que pueden ocurrir en la composición de la leche son:
disminución de la cantidad y la calidad de caseína sintetizada, disminución
de la grasa butirosa, disminución de la lactosa, aumento de
la concentración de sodio, aumento de los cloruros, aumento de las
proteínas del suero sanguíneo, aumento de enzimas y aumento de las
células somáticas. Por lo tanto, si la calidad de la leche no es buena
puede conducir a una disminución en los rindes económicos (8) ya que
se afecta el rendimiento del queso y la vida útil de todos los productos
lácteos (9).
Existen suficientes evidencias que sostienen que las variaciones
individuales en la respuesta inmune, ante la exposición a un agente
patógeno, son consecuencia de factores genéticos y ambientales (10).
Consecuentemente, existe un considerable interés en definir marcadores
genéticos e inmunológicos que puedan ser utilizados para selecciona
animales a favor de la resistencia a mastitis, debido a que es una
de las enfermedades más frecuentes en el ganado bovino lechero y la
que ocasiona las mayores pérdidas económicas (11).
La resistencia genética es un rasgo multigénico, es decir, determinado
por muchos genes, entre los cuales se encuentran los del Complejo
Principal de Histocompatibilidad (MHC). Para desencadenar una respuesta
inmunológica apropiada en los mamíferos, los antígenos extraños,
por ejemplo bacterias, que ingresan en los animales deben ser
fragmentados y presentados a sus respectivos receptores o moléculas
presentadoras de antígeno.
Los loci del MHC constituyen los principales genes candidatos para
el estudio de asociación entre marcadores genéticos y resistencia/susceptibilidad
a enfermedades infecciosas. Cada uno de sus genes a su
vez tiene muchos alelos por lo que es también polimórfico. Este polimorfismo
tiene un profundo efecto en el reconocimiento de los antígenos
por las células T y la combinación de poligenia y polimorfismo
extiende ampliamente el rango de péptidos que pueden ser presentados
a las células T por un individuo (12).
En Bovinos el MHC se denomina Bovine Leukocyte Antigen (BoLA)
y ha sido mapeado en el autosoma bovino 23 (BTA 23) dentro de una
extensión de 4.000 Kb que contiene más de 154 genes estrechamente
ligados (13).
El BoLA está conformado por genes de clase I, II y III y los polimorfismos
de las moléculas de clase II han sido asociados en vacas
lecheras con resistencia/susceptibilidad a enfermedades infecciosas
tales como mastitis clínica, leucosis y neosporosis entre otras (14–18) Las moléculas de clase II se subdividen en IIa y IIb. Los genes y los productos
de clase IIa son los más estudiados porque presentan altos niveles
de polimorfismo. Esta región comprende los grupos de genes DR y DQ.
Se han identificado un total de catorce genes: la subregión DR es la
más conservada a través de las especies y posee un locus DRA, tres loci
DRB, cinco loci DQA (DQA1, DQA2, DQA3, DQA4, DQA5) y 5 loci DQB (19).
Se han reportado hasta el momento un total de 65 alelos DQA y 87
alelos DQB (20).
El BoLA-DQ es menos polimórfico que el BoLA-DR pero no por eso
menos importante ya que se ha demostrado que su función es tan efectiva
como la de las moléculas DR (19,21). Por esta razón, se considera que
las duplicaciones de los genes BoLA-DQA y DQB contribuyen a la diversidad
de moléculas presentadoras de antígenos en el ganado bovino.
Algunos individuos llevan una sola copia de DQA y DQB, mientras
otros tienen haplotipos duplicados. Cerca de la mitad de los haplotipos
de clase II presentan duplicación de los genes DQ (22). Cuando los genes
DQ se encuentran duplicados ambos conjuntos de genes DQ pueden
expresarse (23,24).
Sin embargo, es poco el avance de las investigaciones para conocer
si los loci específicos del BoLA de clase II están individualmente asociados
con bajos y altos CCS como medida de resistencia/susceptibilidad,
o si otros genes ligados son responsables de dicha asociación.
Además, el BoLA parece tener influencia sobre otros rasgos tales como
producción de leche y reproducción (11,25) y a variaciones en la respuesta
inmune individual a antígenos.
En bovinos, diferentes trabajos han reportado asociaciones entre
los alelos de clase II del BoLA y la resistencia/susceptibilidad a enfermedades
infecciosas, tales como el linfoma inducido por el virus
de la leucosis bovina (bovine leukemia virus, BLV) (17,18,26–30) y mastitis
(4,6,11,14,16,26,31–34, 35).
Por lo tanto el objetivo general del presente estudio consistió en
analizar los polimorfismos del gen BoLA-DQA1 y detectar su asociación
con la resistencia / susceptibilidad a mastitis medida a través del
conteo de células somáticas en leche.
El estudio experimental se llevó a cabo en animales de la raza
Holstein de la Provincia de La Pampa.
Se extrajeron muestras de 5 ml de sangre entera a 60 animales
y las mismas se conservaron con anticoagulante a -20 ºC hasta su utilización. Estos individuos se seleccionaron porque presentaban registros
de al menos dos lactancias.
Por otra parte, se registraron mensualmente los conteos de células
somáticas (CCS) y registros de producción tales como litros de leche,
porcentaje de grasa en leche y porcentaje de proteínas a lo largo de
toda la lactancia durante cuatro años. El promedio de controles lecheros
de los individuos seleccionados fue de 35 registros a lo largo de los
4 años de estudio.
DISEÑO EXPERIMENTAL
Los 60 animales se categorizaron en dos grupos: grupo caso y grupo control. El primero representado por individuos con conteos de células somáticas mayor o igual a 250.000 por mililitro leche (CCS ≥250.000 cel/ml). Este grupo incluyó animales que tuvieron mastitis al menos dos veces. El grupo control estaba conformado por individuos con registro de células somáticas menor a 250.000 células por mililitro de leche (CCS <250.000 cel/ml). Se tomó como referencia el límite de 250.000 células por mililitro de leche ya que es un indicador estándar de individuos con mastitis cuando lo superan ó de individuos fisiológicamente normales con menos de 250.000 cel/ml (36). El rodeo analizado en el presente estudio pertenecía a un establecimiento lechero que no poseía registros genealógicos pero sí registros de la procedencia de los animales. Esto último permitió verificar que el rodeo fue armado con animales de distintos tambos, por lo tanto, los animales estudiados no compartían madre o padre.
TIPIFICACIÓN DEL EXÓN 2 DEL GEN BOLA-DQA1 MEDIANTE PCR-RFLP (REACCIÓN EN CADENA DE LA POLIMERASAPOLIMORFISMO DE LONGITUD DE LOS FRAGMENTOS DE RESTRICCIÓN)
El ADN genómico se extrajo a partir de las muestras de sangre entera
mediante el kit DNA Wizard (Promega, USA), según las indicaciones
del proveedor. El gen BoLA-DQA1 se genotipo mediante PCR anidado
(37). Para la primera ronda de amplificación del exón 2 se utilizaron
los oligonucleótidos DQA1-intL2 CACAAATGAAGCCCACAATG y DQA1-
677R CCCTAGGGAAAAGGGAGTGA. Las reacciones se realizaron utilizando
4 μl de ADN molde, 15,35 μl de agua, 2,5 μl de buffer, 2 μl de
dNTPs, 1 μl de cebadores y 0,15 μl de Taq en un volumen final de 25 μl.
El perfil térmico para la primera ronda incluyó una etapa de desnaturalización
inicial a 94ºC durante 2 minutos, 15 ciclos de 94ºC durante 20 segundos, 60ºC durante 20 segundos, 72ºC durante 40 segundos y
para terminar una extensión final de 1 ciclo 72ºC durante 4 minutos.
En la segunda reacción los cebadores utilizados fueron:
DQAintL3 GCCCACAATGTTTGATAGTC y DQA1ex2REV
GGGRACACATACTGTTGGTAG (R= A o G). Las reacciones se realizaron
sobre 5 μl de producto de la primera ronda 33,7 μl de agua, 5 μl de buffer,
4μl de DNTPs, 2μl de cebadores y 0,3 μl de Taq polimerasa.
Las condiciones de amplificación fueron las siguientes: desnaturalización
inicial a 94°C durante 2 minutos, seguido por 30 ciclos de
94°C durante 20 segundos, 60°C durante 20 segundos y 72°C durante
40 segundos, para terminar con una extensión final a 72°C durante 4
minutos.
Con esta reacción se obtuvieron fragmentos de 355 pb que incluyen
el exón 2 y sectores flanqueantes de los intrones 1 y 2 del gen BoLADQA1.
La correcta amplificación se confirmó en un gel de agarosa al
1%.
Los productos de amplificación se digirieron con las enzimas Hae
III, Mbo I, Hha I. Los fragmentos de restricción se resolvieron en geles
de poliacrilamida 8% que se utilizaron para separar las bandas de los
productos de la digestión enzimática (PCR-RFLP).
Luego para la visualización de las mismas los geles se tiñeron con
nitrato de plata.
Los sitios de restricción de distintas enzimas para cada alelo se determinaron
utilizando el software Webcutter2.0 (38) Para cumplir dicho
objetivo se bajaron las secuencias correspondientes a los alelos
del BoLA-DQA1 de la página IPD-MHC Database (20,39).
La clasificación de los alelos se realizó teniendo en cuenta las combinaciones
de los patrones de restricción obtenidos en forma independiente
para las enzimas antes mencionadas.
La lectura de los alelos se basó en la nomenclatura del 5th BoLA
workshop (BoLA Nomenclature, International, Society for Animal
Genetics) (40).
TIPIFICACIÓN DEL EXÓN 2 DEL GEN BOLA-DQA1 POR PCR-SBT (REACCIÓN EN CADENA DE LA POLIMERASA-TIPIFICACIÓN BASADA EN SECUENCIACIÓN)
Para realizar esta técnica se emplearon los mismos cebadores que los utilizados para la tipificación por PCR-RFLP y las condiciones para las dos rondas de amplificación también fueron las mismas. Los amplificados se corrieron en geles de acrilamida al 6% 1X TBE y luego se tiñeron con Bromuro de Etidio. La secuenciación de los productos de PCR se llevó a cabo mediante un secuenciador de tecnología capilar MEGABACE 1000 (GE Healthcare, USA) utilizando el kit DYEnamic ET Terminator Kit (GE Healthcare). Las secuencias crudas fueron editadas usando el programa (Sequence Analyzer (GE Healthcare) (41).
ANÁLISIS ESTADÍSTICO
La estimación de las frecuencias génicas y el número de alelos se realizó por conteo directo. La heterocigosidad observada (ho) y esperada (he) se calcularon de acuerdo a Nei (42) utilizando el programa ARLEQUIN 3.5 (43). Las desviaciones del Equilibrio de Hardy–Weinberg (EHW) se estimaron a través del índice FIS (44), utilizando el test exacto y el test de probabilidad incluidos en el programa GENEPOP 4.0 (45). La significancia del estadístico FIS se calculó mediante el método de Markov Chain (46).
ANÁLISIS DE SECUENCIAS
Las secuencias de ADN se alinearon con el programa CLUSTAL-W 1.8 multiple alignment software (47). La asignación de alelos se realizó mediante la utilización del programa HAPLOFINDER (48).
ESTUDIOS DE ASOCIACIÓN ENTRE EL CCS Y LA FRECUENCIA GÉNICA DEL BOLA-DQA1
Se realizaron mediante el test exacto de Fisher, el de Odds Ratio (OR) de Woolf-Haldane y los intervalos de confianza (CI). Estos cálculos se llevaron a cabo mediante el paquete de R Epitools (49).
CARACTERIZACIÓN DE LOS POLIMORFISMOS PRESENTES DEL GEN BOLA-DQA1
Se genotipó el exón 2 del BoLA-DQA1 por PCR-RFLP en 60 animales y de esta manera se identificaron un total de 16 alelos. En la tabla 1 se pueden observar los resultados obtenidos de heterocigosidad esperada y observada, como así también las frecuencias génicas de los dos grupos estudiados: el grupo caso (CCS ≥250.000 cel/ml) y el grupo control (CCS <250.000 cel/ml).
Tabla 1. Frecuencias génicas de los alelos definidos por PCR-RFLP para el gen
BoLA-DQA1 en la población Holstein estudiada
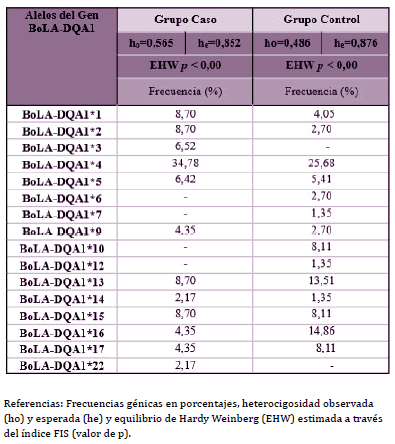
Veintitrés de los animales analizados por PCR-RFLP fueron nuevamente tipificados mediante la técnica de PCR-SBT para confirmar los alelos detectados. Se encontraron un total de 8 alelos por medio de PCR-SBT y en todos los casos los resultados fueron coincidentes con los resultados obtenidos por PCR-RFLP. Estos alelos fueron: BoLADQA1* 0204, *10011, *10012, *12012, *0102, *1203, *12021 y *0101.
ASOCIACIÓN DEL GEN BOLA-DQA1 CON EL NÚMERO DE CÉLULAS SOMÁTICAS
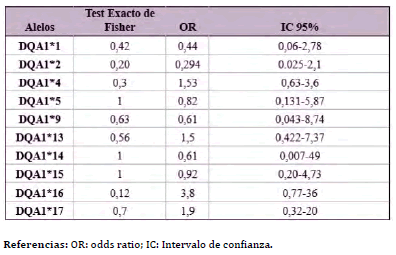
El test exacto de Fisher y el Odds Ratio (OR) de Woolf- Haldane también fueron utilizados para estudiar la asociación entre el CCS y los alelos del gen BoLA-DQA1 genotipados por PCR-RFLP (Tabla 2). Ninguno de los valores de OR calculados fueron significativos (p > 0,12).
Tabla 2. Asociación de alelos del BoLA-DQA1, definidos por PCR-RFLP con el
conteo de células somáticas (CCS) en la población Holstein estudiada
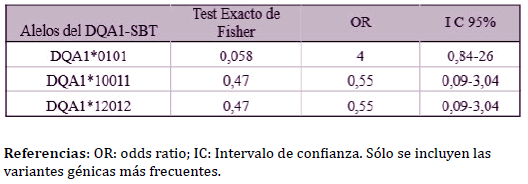
También se estudió la asociación entre el CCS y los alelos del gen
BoLA-DQA1 genotipados por PCR-SBT. Los valores de OR sólo se calcularon
para aquellos alelos con frecuencias mayores o iguales al 15%,
dado que el resto presentaron valores bajos de frecuencia génica.
Si bien el alelo BoLA-DQA1*0101, correspondiente al alelo BoLADQA1*
16 tipificado por PCR-RFLP, fue el mayoritario en el grupo con
bajo CCS, evidenció un valor de OR de 4 (p = 0,058), lo que indicaría
una posible asociación entre dicho alelo y susceptibilidad a padecer
la enfermedad (Tabla 3). Cabe destacar que el alelo del BoLA-DQA1*16
(PCR-RFLP) también mostró un valor de riesgo cercano a 4, aunque en
este caso la probabilidad no fue significativa (p= 0,12) (Tabla 2).
En cuanto a los alelos BoLA-DQA1*10012 y BoLA-DQA1*10011, que
fueron los más frecuentes en el grupo con alto CCS con frecuencia génica del 25%, presentaron valores de OR no significativos e inferiores
a 1.
Tabla 3. Asociación de los alelos BoLA-DQA1, definidos por PCR-SBT, con conteo
de células somáticas (CCS) en la población Holstein estudiada
En el presente trabajo se caracterizaron los polimorfismos presentes
en el exón 2 del gen BoLA-DQA1 en bovinos de raza Holstein de La
Pampa, detectándose que ambos grupos analizados (caso y control)
tenían como alelo más frecuente a la variante BoLA-DQA1*4 y a la menos
frecuente al BoLA-DQA1*14 es decir se encuentran distribuídos de
manera similar en ambos grupos del rodeo de raza Holstein analizado.
Este resultado coincide con lo reportado por Miyasaka et al. (50) quienes
analizaron rodeos de distintas razas bovinas de Japón.
En nuestros resultados se evidenció un aumento significativo de
individuos homocigotas. El rodeo analizado no poseía registros genealógicos
pero sí registros de la procedencia de los animales, esto
permitió verificar que el rodeo fue armado con animales de distintos
tambos. Pero no se descarta cierto grado de consanguinidad entre los
animales del mismo tambo ocasionado por la propia dinámica de la
cría, pudiendo existir estructuración poblacional. Como los valores de
ho dependen de las frecuencias génicas de origen de las muestras,
se explicaría el aumento de individuos homocigotas. Takeshima et al. (51) y Miyasaka et al. (50), propusieron que los individuos heterocigotos
para el gen BoLA-DQA1 presentan ventajas contra enfermedades
infecciosas provocadas por determinados patógenos, pero aún no se
conoce claramente cuáles son los mecanismos de selección que favorecen
a los genotipos heterocigotos. El aumento de la heterocigosidad
se corresponde con el incremento de la resistencia a enfermedades
infecciosas ya que favorece la diversidad de las células encargadas de presentar antígenos a los linfocitos T, generando de esta manera un
diverso repertorio de éstas. Así por ejemplo, estos autores reportaron
que el genotipo BoLA-DQA1*0101/*0101 estaba asociado a susceptibilidad
a mastitis causada por Streptococcus sp. y el genotipo BoLADQA1*
10011/*10011 con susceptibilidad a mastitis provocada por
Escherichia coli.
Una submuestra de animales a los que se tipificó el exón 2 del gen
BoLA-DQA1 por PCR-RFLP, fueron también genotipados por PCR-SBT.
El alelo BoLA-DQA1*0101 fue el más frecuente, en coincidencia con lo
observado por Takeshima et al.(51) en una población Holstein de Japón,
y por Misayaka et al.(50) en ganado Japonés Negro y Holstein. Kulaj et
al. (52) encontraron resultados similares en una población de ganado
Holstein Friesian (variedad negra y blanca) de Polonia. El alelo BoLADQA1*
10012 fue el segundo alelo más abundante coincidiendo con lo
reportado por Miyasaka et al. (50) en la raza Japonesa Negra. Takeshima
et al. (51) encontraron que el alelo BoLA-DQA1*10012 se encontraba
en una frecuencia moderada (alrededor del 10%) en la población de
Holstein que analizaron. En tanto, Kulaj et al. (52) lo encontraron en una
frecuencia del 2,82% en ganado Holstein de la población estudiada.
Por otro lado, en el grupo con alto CCS analizado en el presente estudio
los alelos BoLA-DQA1*10011 y *10012 (DQA1*4) fueron los más
frecuentes.
El alelo BoLA-DQA1*12021 fue el más frecuente en el ganado Jersey
(0,336), el alelo BoLA-DQA1*10012, fue el alelo más frecuente en el
ganado negro japonés (0,332) y el alelo BoLA-DQA1*0204 lo fue en el
Shorthorn japonés (0,23) según lo reportado por Takeshima et al. (51).
Se encontraron resultados similares en tres rebaños de ganado negro
japonés, en los que el alelo BoLA-DQA1*10012 tenía una frecuencia
de 0,32 a 0,42 (50).
Se calcularon los OR para los alelos BoLA-DQA1 tipificados por PCRRFLP
pero ninguno evidenció una asociación significativa con resistencia/
susceptibilidad a mastitis medida a través del CCS. En el caso
de los alelos genotipados mediante PCR-SBT el alelo BoLA-DQA1*0101
presentó un valor de OR de 4 (p = 0,058), lo cual indicaría una tendencia
a la asociación entre dicho alelo y la mastitis. Este resultado coincide
con lo reportado por Takeshima et al. (51) quienes asocian esta variante
con la susceptibilidad a mastitis causada por Streptococcus sp.
En el caso de los alelos BoLA-DQA1*10012 y *10011, los resultados
obtenidos no evidenciaron valores significativos de OR. Por el contrario,
Takeshima et al.(51) reportaron una asociación significativa entre
el alelo BoLA-DQA1*10011 y mastitis provocada por Escherichia coli. En recientes trabajos realizados por Takeshima et al. (53), compararon
alelos del gen BoLA-DQA1 y BoLA-DRB3 para estudiar la carga proviral
del virus de leucosis y encontraron una menor asociación en el
marcador DQA1 que con el DRB3 en vacas Holstein de Japón (OR = 11,5
para el gen BoLA-DQA1; OR = 13,9 para BoLA-DRB3 y OR = 6,2 para los
haplotipos de clase II del BoLA).
De acuerdo a los resultados obtenidos y comparados con la bibliografía
disponible, la distribución de las frecuencias génicas del locus
BoLA-DQA1 difiere significativamente según la raza de ganado estudiada.
En nuestro análisis, realizado en animales de la raza Holstein, el
alelo BoLA-DQA1*0101, correspondiente al alelo BoLA-DQA1*16 tipificado
por PCR-RFLP, evidenció una posible asociación entre dicho alelo
y la susceptibilidad a padecer la mastitis.
Es importante destacar que diferentes patógenos están presentes
en el medio ambiente de cada rodeo y el mismo alelo puede responder
de manera diferente para cada uno de ellos. También pudieron estar
comprometidos factores ambientales que involucran el manejo, el estado
sanitario y la alimentación del rodeo en estudio.
Los estudios de asociación entre los genes del BoLA y mastitis basados
en marcadores moleculares podrían constituir una efectiva herramienta
para el control de enfermedades infecciosas, ya sea mediante
su aplicación en los programas de selección genética o mediante el desarrollo
de vacunas y, de esta forma, reducir los costos sanitarios que
afectan a la producción lechera.
La resistencia/susceptibilidad en vacas con mastitis podría variar
dependiendo de la combinación inter e intra alélica de los diferentes
loci del BoLA.
En próximos estudios sería conveniente la genotipificación de otros
genes candidatos para poder calcular con certeza la asociación entre
las moléculas del BoLA con resistencia/susceptibilidad a enfermedades
multifactoriales como la mastitis, mediante la aplicación de las
nuevas técnicas, especialmente aquellas basadas en las tecnologías de
NGS (Next Generation Sequencing), y así incluir el análisis simultáneo
de los diferentes loci involucrados en la resistencia susceptibilidad a
enfermedades infecciosas en vacas lecheras.
1. Quevedo W. Recuento de células somáticas (rsc), como indicador en la resistencia de la mastitis bovina. Revista Ciencia, Tecnología e Innovación [Internet]. 2018; Available from: http://www.scielo.org.bo/scielo. php?pid=S2225-87872018000100005&script=sci_arttext
2. Kerr DE, Wellnitz O. Mammary expression of new genes to combat mastitis. J Anim Sci. 2003; 81 Suppl 3:38–47.
3. Blum SE, Heller ED, Leitner G. Long term effects of Escherichia coli mastitis. Vet J. 2014 Jul; 201(1):72–7.
4. Sharif S, Mallard BA, Wilkie BN, Sargeant JM, Scott HM, Dekkers JC, et al. Associations of the bovine major histocompatibility complex DRB3 (BoLA-DRB3) alleles with occurrence of disease and milk somatic cell score in Canadian dairy cattle. Anim Genet. 1998 Jun; 29(3):185–93.
5. Ariznabarreta A, Gonzalo C, San Primitivo F. Microbiological quality and somatic cell count of ewe milk with special reference to staphylococci. J Dairy Sci. 2002 Jun; 85(6):1370–5.
6. Park YH, Joo YS, Park JY, Moon JS, Kim SH, Kwon NH, et al. Characterization of lymphocyte subpopulations and major histocompatibility complex haplotypes of mastitis-resistant and susceptible cows. J Vet Sci. 2004 Mar; 5(1):29–39.
7. Bochantin K, Bewley JM. 0042 The importance of mastitis management practices in maintaining milk quality in the United States. J Anim Sci. 2016; 94(suppl_5):19–20.
8. Guimarães JLB, Brito MAV, Lange CC, Silva MR, Ribeiro JB, Mendonça LC, et al. Estimate of the economic impact of mastitis: A case study in a Holstein dairy herd under tropical conditions [Internet]. Vol. 142, Preventive Veterinary Medicine. 2017. p. 46–50. Available from: http://dx.doi.org/10.1016/j.prevetmed.2017.04.011
9. Bedolla CC, de León MP. Pérdidas económicas ocasionadas por la mastitis bovina en la industria lechera. REDVET - Revista Electrónica de Veterinaria. 2008; 9(4):1–26.
10. Ting JP-Y, Trowsdale J. Genetic control of MHC class II expression. Cell. 2002 Apr; 109 Suppl: S21–33.
11. Rupp R, Hernandez A, Mallard BA. Association of bovine leukocyte antigen (BoLA) DRB3.2 with immune response, mastitis, and production and type traits in Canadian Holsteins. J Dairy Sci. 2007 Feb;90(2):1029–38.
12. Brown JH, Jardetzky TS, Gorga JC, Stern LJ, Urban RG, Strominger JL, et al. Three-dimensional structure of the human class II histocompatibility antigen HLA-DR1. Nature. 1993;364:33–9. Available from: http://dx.doi.org/10.1038/364033a0
13. Tellam RL, Lemay DG, Van Tassell CP, Lewin HA, Worley KC, Elsik CG. Unlocking the bovine genome. BMC Genomics. 2009;10:193. Available from: http://dx.doi. org/10.1186/1471-2164-10-193
14. Takeshima S-N, Aida Y. Structure, function and disease susceptibility of the bovine major histocompatibility complex. Anim Sci J. 2006 Apr; 77(2):138–50.
15. Schwab AE, Geary TG, Baillargeon P, Schwab AJ, Fecteau G. Association of BoLA DRB3 and DQA1 alleles susceptible to Neospora caninum and reproductive outcome in Quebec Holstein cattle. Vet Parasitol. 2009 Oct 28; 165(1):136–40.
16. Baltian LR, Ripoli MV, Sanfilippo S, Takeshima SN, G. G. Association between Bo- LA-DRB3 and somatic cell count in Holstein cattle from Argentina. Mol Biol Rep. 2012 39 (7):7215-7220 Available from: https://link.springer.com/article/10.1007/ s11033-012-1526-y
17. Baltian LR, Follmer AV, Peratta DL, Schmidt EE, Severini RA, Borrego C, et al. Polimorfismos del exón 2 del gen BoLA-DRB3 asociados con resistencia/susceptibilidad a leucosis en ganado Holstein de La Pampa/Polymorphisms of BoLA-DRB3 gene and its association with resistance/susceptibility to Leukosis in Holstein cattle from La. Ciencia Veterinaria. 2016; 18(1):9–27.
18. Polat M, Moe HH, Shimogiri T, Moe KK, Takeshima S-N, Aida Y. The molecular epidemiological study of bovine leukemia virus infection in Myanmar cattle. Arch Virol. 2017 Feb; 162(2):425–37.
19. Miyasaka T, Takeshima SN, Sentsui H, Aida Y. Identification and diversity of bovine major histocompatibility complex class II haplotypes in Japanese Black and Holstein cattle in Japan. J Dairy Sci. 2012;95 (1):420-431 Available from: https://www.sciencedirect com/science/article/pii/S0022030211007132
20. EBI Web Services. IPD-MHC Database [Internet]. [cited 2021 May 12]. Available from: https://www.ebi.ac.uk/ipd/mhc/
21. Glass EJ, Oliver RA, Russell GC. Duplicated DQ haplotypes increase the complexity of restriction element usage in cattle. J Immunol. 2000 Jul 1; 165(1):134–138.
22. Gelhaus A, Förster B, Wippern C, Horstmann RD. Evidence for an additional cattle DQA locus, BoLA-DQA5. Immunogenetics. 1999 Apr;49(4):321–7.
23. Russell GC, Gallagher A, Craigmile S, Glass EJ. Characterization of cattle cDNA sequences from two DQA loci. Immunogenetics. 1997;45(6):455–8.
24. Takeshima S, Aida Y. Polymorphism and disease resistance of bovine major histocompatibility complex. J Animal Gen. 2007;35:51–64. Available from: http://dx.doi. org/10.5924/abgri2000.35.51
25. Ashwell MS, Heyen DW, Sonstegard TS, Van Tassell CP, Da Y, VanRaden PM, et al. Detection of Quantitative Trait Loci Affecting Milk Production, Health, and Reproductive Traits in Holstein Cattle. J Dairy Sc. 2004;87:468–75. Available from: http://dx.doi. org/10.3168/jds.s0022-0302(04)73186-0
26. AIDA, Y. Influence of host genetic differences on leukemogenesis induced bovine leukemia virus. AIDS Res Hum Retroviruses. 2001; 17:S12.
27. Burbano M, Toro R, Montoya F, Ariza F, Tobón JI, Gallego J, et al. Caracterización del locus BoLA-DRB3 en ganado criollo colombiano y asociación con resistencia a enfermedades. Arch Zootec. 2005; 54(206):349–56.
28. Martinez ML, Machado MA, Nascimento CS, Silva MVGB, Teodoro RL, Furlong J, et al. Association of BoLA-DRB3.2 alleles with tick (Boophilus microplus) resistance in cattle. Genet Mol Res. 2006 Aug 31; 5(3):513–24.
29. Juliarena MA, Poli M, Sala L, Ceriani C, Gutierrez S, Dolcini G, et al. Association of BLV infection profiles with alleles of the BoLA-DRB3.2 gene. Anim Genet. 2008 Aug; 39(4):432–8.
30. Panei CJ, Suzuki K, Echeverria MG, Serena MS, Metz GE, Gonzalez ET. Association of Bo- LA-DRB3.2 Alleles with Resistance and Susceptibility to Persistent Lymphocytosis in BLV Infected Cattle in Argentina. Int J Dairy Sci. 2009;4:123–8. Available from: http:// dx.doi.org/10.3923/ijds.2009.123.128
31. Dietz A B, Detilleux JC, Freeman A E, Kelley D H, Stabel JR, Kehrli M E. Genetic association of bovina lymphocyte antigen DRB3 alleles with immunological traits of Holstein cattle. J Dairy Sci. 1997; (80):400–5.
32. Kelm SC, Detilleux JC, Freeman AE, Kehrli ME Jr, Dietz AB, Fox LK, et al. Genetic association between parameters of innate immunity and measures of mastitis in periparturient Holstein cattle. J Dairy Sci. 1997 Aug; 80(8):1767–75.
33. Zambrano JC., Echeverri JZ., López-Herrera A. Alelos del gen BoLA DRB3.2 están asociados con mastitis en vacas lechera. Rev Colom Cienc Pecu. 2011; 24(2):145–56.
34. Baltian LR., Rípoli MV., Giovambattista G. Determination of amino acid motifs present in the antigen-binding site of BoLA-DRB3 alleles in a Holstein population of La Pampa y su asociación con mastitis. Ciencia Veterinaria. 2014; 16: 9–27. Available from:http://dx.doi.org/10.19137/cienvet2014-1611
35. Chu MX, Ye SC, Qiao L, Wang JX, Feng T, Huang DW, et al. Polymorphism of exon 2 of BoLA-DRB3 gene and its relationship with somatic cell score in Beijing Holstein cows. Mol Biol Rep. 2012 Mar;39(3):2909–14.
36. National Mastitis Council - National Mastitis Council [Internet]. 2016 [cited 2021 May 15]. Available from: http://nmconline.org.
37. Takeshima S, Miki A, Kado M, Aida Y. Establishment of a sequence-based typing system for BoLA-DQA1 exon 2. Tissue Antigens. 2007 Feb;69(2):189–99.
38. WebCutter [Internet]. WebCutter. Available from: http://heimanlab.com/cut2.html
39. EMBL-EBI. The European Bioinformatics Institute < EMBL-EBI [Internet]. [cited 2021 May 14]. Available from: http://www.ebi.ac.uk/
40. Russell GC, Davies CJ, Andersson L, Mikko S, Ellis SA, Hensen EJ, et al. BoLA class II nucleotide sequences, 1996: report of the ISAG BoLA Nomenclature Committee. Animal Genetics. 1997;28:169–80. Available from: http://dx.doi. org/10.1111/j.1365-2052.1997.00107.x
41. Daum C, Tighe D, Philip L, Mihalkanin D, Spurrell C, Miller D, et al. The Joining of Competitors: The Dual Operation of the ABI 3730xl and GE MegaBACE4500 DNA Sequence Analyzers at the DOE Joint Genome Institute. 2006 Feb 6 [cited 2021 May 16]; Available from: https://escholarship.org/uc/item/9hq0x9zp
42. Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics. 1978 Jul; 89(3):583–90.
43. Eindows. Mol Ecol Resour. 2010;10:564– 7. Available from: http://dx.doi.org/10.1111/j.1755-0998.2010.02847.xxcoffier L, Lischer HEL. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and W
44. Weir BS, Cockerham CC. ESTIMATING F-STATISTICS FOR THE ANALYSIS OF POPULATION STRUCTURE. Evolution. 1984 Nov; 38(6):1358–70.
45. Rousset F. genepop’007: a complete re-implementation of the genepop software for Windows and Linux. Mol Ecol Resour. 2008 Jan; 8(1):103–6.
46. Guo SW, Thompson EA. Performing the exact test of Hardy-Weinberg proportion for multiple alleles. Biometrics. 1992 Jun;48(2):361–72.
47. Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Res. 1994 Nov 11;22(22):4673–80.
48. Miltiadou D, Law AS, Russell GC. Establishment of a sequence-based typing system for BoLA-DRB3 exon 2. Tissue Antigens. 2003 Jul;62(1):55–65.
49. Ripley BD. The R project in statistical computing. MSOR Connections The newsletter of the LTSN Maths [Internet]. 2001; Available from: https://citeseerx.ist.psu.edu/ viewdoc/download?doi=10.1.1.449.6899&rep=rep1&type=pdf
50. Miyasaka T, Takeshima S-N, Matsumoto Y, Kobayashi N, Matsuhashi T, Miyazaki Y, et al. The diversity of bovine MHC class II DRB3 and DQA1 alleles in different herds of Japanese Black and Holstein cattle in Japan. Gene. 2011 Feb 1;472(1-2):42–9.
51. Takeshima S, Chen S, Miki M, Kado M, Aida Y. Distribution and origin of bovine major histocompatibility complex class II DQA1 genes in Japan. Tissue Antigens. 2008;72:195– 205. Available from: http://dx.doi.org/10.1111/j.1399-0039.2008.01092.x
52. Kułaj D, Pokorska J, Ormian M, Dusza M. Short Communication: New alleles at the Bo- LA-DQA1 locus in Holstein–Fresian cattle. Can J Anim Sci. 2015 Jun;95(2):161–4.
53. Takeshima S-N, Ohno A, Aida Y. Bovine leukemia virus proviral load is more strongly associated with bovine major histocompatibility complex class II DRB3 polymorphism than with DQA1 polymorphism in Holstein. Retrovirology, 2019; 16 (1): p. 1-6. Available from: https://retrovirology.biomedcentral.com/articles/10.1186/ s12977-019-0476-z
Fecha de recepción: 01/08/2021
Fecha de aceptación para su publicación: 01/11/2021