DOI: http://dx.doi.org/10.19137/cienvet202325101
![]() Esta obra se publica bajo licencia Creative Commons 4.0 Internacional. (Atribución-No Comercial-
Compartir Igual) a menos que se indique lo contrario, http://www.creativecommons.org.ar/licencias.html
Esta obra se publica bajo licencia Creative Commons 4.0 Internacional. (Atribución-No Comercial-
Compartir Igual) a menos que se indique lo contrario, http://www.creativecommons.org.ar/licencias.html
ARTÍCULOS DE INVESTIGACIÓN
Evaluación de la Sinergia de la Actividad
Antimicrobiana de Cepas de Bacterias Lácticas
Contra Patógenos
Antimicrobial Sinergy Evaluation of Lactic
Bacteria Strains Against Pathogens
Avaliação da Sinergia da Atividade
Antimicrobiana de Cepas de Bactérias Láticas
Contra Patógenos
Pino AI 1, Albarracín L. 2, Parra N 3, Toledo JR 3, Villena J 2, Quilodrán-Vega S1
1 Laboratorio de Microbiología de Alimentos, Departamento de Patología y Medicina
Preventiva, Facultad de Ciencias Veterinarias, Universidad de Concepción, Chillán,
Chile.
2 Laboratorio de Inmunobiotecnología, Centro de Referencia para Lactobacilos
(CERELA-CONICET), Tucumán, Argentina.
3 Laboratorio de Biotecnología y Biofármacos, Departamento de Fisiopatología,
Facultad de Ciencias Biológicas, Universidad de Concepción, Concepción, Chile.
Correo electrónico: squilodran@udec.cl
RESUMEN Una de las propiedades biológicas más notables atribuidas a los probióticos es su actividad antimicrobiana. La investigación sobre probióticos se ha centrado tradicionalmente en cepas individuales, sin embargo, las combinaciones de cepas probióticas pueden tener un espectro de acción más amplio o efectos sinérgicos, potenciando su acción protectora. El objetivo de esta investigación fue evaluar efecto sinérgico de cepas de bacterias lácticas aisladas de leche materna de diferentes orígenes sobre la inhibición de patógenos bacterianos de importancia en medicina humana y animal. Se emplearon combinaciones de siete cepas de bacterias lácticas juntadas en pares y se estudió su efecto inhibitorio sobre los patógenos bacterianos Escherichia coli ATCC 25922, Salmonella enterica ATCC 13076, Klebsiella pneumoniae ATCC 13883, y Acinetobacter baumannii ATCC 19606 y una cepa de E. coli enterohemorrágica. La combinación de las cepas TUCO-5E + TUCO-L2 fue la más efectiva en la inhibición del desarrollo de todos los patógenos evaluados, destacando su efecto sobre K. pneumoniae y A. baumannii. Cinco de las combinaciones evaluadas mostraron efectos sinérgicos en la inhibición de patógenos extraintestinales: a) se observó efecto sinérgico entre las cepas TUCO-5E + TUCO-L2 para la inhibición de K. pneumoniae y A. baumannii, b) efecto sinérgico para TUCO-5E + TUCO-2, TUCO-5 + TUCO-2, y TUCO-3 + TUCO-17 sobre K. pneumoniae y, c) efecto sinérgico para TUCO-16 + TUCO-17 sobre A. baumannii. Las combinaciones que mostraron sinergia en el efecto antimicrobiano son buenas candidatas para el desarrollo de probióticos multicepa para la prevención y el tratamiento de infecciones bacterianas específicas.
Palabras clave: Probióticos; Sinergia; Lactobacillus; Actividad Antimicrobiana
ABSTRACT One of the most notable biological properties attributed to probiotics is their antimicrobial activity. Probiotics research has traditionally been focused on single strains, however, probiotic strains combinations can have a broader spectrum of action or synergistic effects, enhancing their protective action. The objective of this research was to evaluate the synergistic effect of lactic acid bacteria isolated from breast milk of different origins on the inhibition of bacterial pathogens of importance in human and animal medicine. Combinations of seven strains of lactic acid bacteria paired together were used and their inhibitory effect on bacterial pathogens Escherichia coli ATCC 25922, Salmonella enterica ATCC 13076, Klebsiella pneumoniae ATCC 13883, Acinetobacter baumannii ATCC 19606 and an enterohemorragic E. coli strain was investigated. The combination TUCO-5E + TUCO-L2 was the most effective in inhibiting the growth of all pathogens evaluated, highlighting its effect on K. pneumoniae y A. baumannii. Five of the evaluated combinations showed synergistic effects in the inhibition of extraintestinal pathogens: a) a synergistic effect was observed between strains TUCO-5E + TUCO-L2 for the inhibition of K. pneumoniae and A. baumannii, b) synergistic effect for TUCO-5E + TUCO-2, TUCO-5 + TUCO-2, and TUCO-3 + TUCO-17 on K. pneumoniae and, c) synergistic effect for TUCO-16 + TUCO-17 on A. baumannii. The combinations that showed synergy in the antimicrobial effect are good candidates for the development of multistrain probiotics for the prevention and treatment of specific bacterial infections.
Keywords: Probiotics; Synergy; Lactobacillus; Antimicrobial Activity
RESUMO Uma das propiedades biológicas mais notáveis atribuídas aos probióticos é a sua atividade antimicrobiana. A investigação sobre probióticos está focado tradicionalmente em cepas individuais, porém, as combinações de cepas probióticas podem ter um espectro de ação mais amplo, ou efeitos de sinergia que potenciam sua ação de proteção. O objetivo desta investigação foi avaliar o efeito de sinergia de cepas de bacterias láticas, isoladas do leite materno de diferentes origens, na inibição de patógenos bacterianos importantes na medicina humana e animal. Sete combinações de cepas de pares de bacterias láticas foram usadas e o efeito de inibição contra patógenos bacterianos Escherichia coli ATCC 25922, Salmonella enterica ATCC 13076, Klebsiella pneumoniae ATCC 13883, e Acinetobacter baumannii ATCC 19606 e uma cepa de E. coli enterohemorrágica. A combinação das cepas TUCO-5E + TUCO-L2 foi a mais efetiva na inibição do crescimento de todos os patógenos avaliados, destacando o efeito contra K. pneumoniae e A. baumannii. Cinco combinações apresentaram efeito de sinergia na inibição de patógenos extraintestinais: a) efeito de sinergia das cepas TUCO-5E + TUCO-L2 na inibição de K. pneumoniae e A. baumannii, b) efeito de sinergia das cepas TUCO-5E + TUCO-2, TUCO-5 + TUCO-2, e TUCO-3 + TUCO-17 contra K. pneumoniae e c) efeito de sinergia das cepas TUCO-16 + TUCO-17 contra A. baumannii. As combinações das cepas que apresentaram sinergia na inibição contra as cepas patógenas são boas candidatas para a elaboração de productos probióticos multicepa para prevenir e tratar infecções bacterianas específicas.
Palavras-chave: Probióticos ; Sinergia; Lactobacillus; Atividade Antimicrobiana
Introducción
Los antibióticos están entre los más destacados avances en
medicina y han traído grandes beneficios al tratamiento y control
de enfermedades infecciosas. Sin embargo, la habilidad de los
microorganismos de evolucionar rápidamente junto al uso
excesivo e inapropiado de los antibióticos han conducido a la
expansión de la resistencia antimicrobiana en las bacterias
patógenas (1,2). La resistencia a los antimicrobianos es una amenaza
a la salud humana y animal, con grandes pérdidas económicas,
fallas en los tratamientos y aumento en la morbilidad y mortalidad.
Esto hace indispensable la reducción del uso de antimicrobianos y
la búsqueda de nuevas opciones preventivas y terapéuticas para el
control de las infecciones(2).
Una de las alternativas al uso de antibióticos son los probióticos (2),
los cuales se definen como microorganismos vivos que
administrados en cantidades adecuadas brindan un beneficio a la
salud del hospedador (3,4).Un microorganismo probiótico debe estar
identificado a nivel de cepa, estar suficientemente caracterizado en
términos de su inocuidad y estabilidad genética, y poseer un
beneficio para la salud respaldado por estudios clínicos en el
huésped en que se pretende usar(5,6,7). Además, los
microorganismos probióticos deben permanecer viables en el
producto en el que se incorporan y mantener una dosis eficaz
durante toda la vida útil del producto(6).
Los probióticos pueden tener diversos efectos beneficiosos para la
salud, por ejemplo, a nivel digestivo, inmunológico, cardiovascular
y neurológico(1,4). El consenso general es que los beneficios para la salud inducidos por los probióticos son cepa específicos y no se
pueden extrapolar a otras cepas, ni siquiera de la misma especie,
sin estudios que lo respalden (1,3) . El efecto más reconocido de los
probióticos y su principal aplicación, tanto en las personas como
en los animales, es la prevención y tratamiento de infecciones
gastrointestinales y diarrea(3,8,9). En medicina humana y veterinaria
se ha documentado que el consumo de probióticos disminuye el
riego de enfermedades infecciosas, y también reduce la duración
media de los síntomas en cuadros agudos comunes, reduciendo de
este modo la necesidad de usar antibióticos (1,10).
Los mecanismos a través de los cuales los probióticos pueden
mejorar la resistencia a infecciones son variados e incluyen la
regulación de la microbiota intestinal, la inhibición de
microorganismos patógenos, y la modulación de la respuesta
inmune (1,4,11). Los probióticos pueden contribuir en la eliminación
de microorganismos patógenos mediante la mejora de la barrera
mucosa, al estimular la producción de mucus, defensinas, IgA y
reforzar uniones intercelulares. También pueden ejercer la
exclusión competitiva de patógenos al utilizar los sitios de unión y
los nutrientes disponibles, o a través de la producción de
sustancias con efecto antimicrobiano como ácidos orgánicos,
peróxido de hidrogeno, óxido nítrico y bacteriocinas (1,4,12). Se ha
demostrado la efectividad de algunas cepas probióticas en la
inhibición de bacterias patógenas gastrointestinales como
Salmonella spp., Escherichia coli, Campylobacter jejuni, Shigella
spp., Helicobacter pylori(11-13).
Los principales orígenes de aislamiento de los microorganismos
probióticos son el tracto digestivo humano o animal, alimentos de
origen animal o vegetal, fermentados o no fermentados, y leche
materna humana o animal(14,15,16). Diversas cepas aisladas desde
leche animal han mostrado características probióticas
prometedoras, como la capacidad de sobrevivir al tránsito
gastrointestinal, la adherencia a la mucosa intestinal,
inmunomodulación y actividad inhibitoria de patógenos. Se han
evaluado cepas de leche de cabra(17,,18), leche de cerda(19,20), leche de
perra(21,22), leche de vaca(18) y leche de llama(23), entre otras.
Frecuentemente los probióticos se estudian como cepas
individuales y luego se formulan comercialmente como productos
multicepa(24), para lograr efectos aditivos o sinérgicos(25). Algunos efectos sinérgicos propuestos son el aumento en la adherencia al
mucus intestinal y el incremento de la actividad antimicrobiana,
aunque también se han registrado efectos negativos como el
antagonismo entre las cepas combinadas(25,26). Existe evidencia de
la efectividad de los probióticos multicepa y multiespecie, tanto en
animales como humanos, por ejemplo, en problemas digestivos,
infecciones respiratorias, e inmunomodulación(13,26,27-29). Sin
embargo, pocos estudios comparan probióticos multicepa y
multiespecie con cepas individuales para evaluar si hay diferencia
en su efecto. Se ha reportado que algunas combinaciones
probióticas tienen mejor adherencia al mucus intestinal que cada
cepa individualmente(30), o mayor inhibición de la adherencia de
patógenos a la mucosa(31,32). En otros estudios hay resultados
variables, donde solo algunas de las combinaciones probióticas
muestran mayor efectividad que las cepas individuales, y en
algunos casos las dosis utilizadas no son equivalentes para la
combinación y la cepa individual, por lo que no se puede concluir
que el efecto sea debido a la sinergia de las cepas y no a una mayor
concentración total de las bacterias probióticas(25,27).
Este trabajo se enfocó en la búsqueda de nuevas combinaciones de
cepas de lactobacilos potencialmente probióticos que sean capaces
de inhibir patógenos bacterianos de importancia en medicina
humana y animal, para su potencial uso en la prevención y
tratamiento de enfermedades infecciosas. Para ello, se evaluó la
actividad antimicrobiana in vitro de las cepas de lactobacilos en
diferentes combinaciones, con el objetivo de encontrar aquellas
que fueran sinérgicas en la inhibición de patógenos, y que tengan
mayor probabilidad de ser efectivas clínicamente.
Las cepas de bacterias lácticas utilizadas pertenecen a la colección
de cultivos del Laboratorio de Microbiología de los Alimentos,
Facultad de Ciencias Veterinarias, Universidad de Concepción
(Chillán, Chile). Las cepas fueron previamente aisladas desde leche
materna de diferentes animales y caracterizadas para su
resistencia a pH 3, y a diferentes concentraciones de Oxgall y NaCl. Levilactobacillus brevis TUCO-5E(20) (leche de cerda), Ligilactobacillus salivarius TUCO-L2(23) (leche de llama), Lactiplantibacillus plantarum TUCO-2 (leche de cabra), Pediococcus pentosaceus TUCO-3 (leche de gata), Lactiplantibacillus plantarum TUCO-5 (leche de cerda), Lactobacillus sp. TUCO-16 (calostro de
perra), y TUCO-17 (leche de perra). Se empleó además la cepaLacticaseibacillus casei Shirota como control.
Para lo experimentos se emplearon además las cepas Escherichia
coli ATCC 25922, aislado de patotipo de E. coli enterohemorrágica
(EHEC), Salmonella enterica ATCC 13076, Klebsiella pneumoniae ATCC 13883, y Acinetobacter baumannii ATCC 19606.
Se evaluaron seis combinaciones de bacterias lácticas, cada una
compuesta por dos cepas potencialmente probióticas: TUCO-5E +
TUCO-L2, TUCO-5E + TUCO-2, TUCO-5 + TUCO-2, TUCO-3 + TUCO-
16, TUCO-3 + TUCO-17, y TUCO-16 + TUCO-17.
El efecto inhibitorio fue evaluado mediante la técnica spot soft
agar(33,34), también llamada spot on lawn(35,36), adaptada con algunas
modificaciones. Las cepas de bacterias lácticas fueron sembradas
en agar MRS (DE MAN, ROGOSA y SHARPE, Oxoid) y fueron
incubadas a 37ºC, en microaerofilia, durante 48 h. Fueron
traspasadas a caldo MRS e incubadas en las mismas condiciones.
Un mililitro de cada cultivo se centrifugó a 3000 rpm por 5
minutos para recolectar el pellet, se hicieron dos lavados con
buffer y se resuspendió en buffer ajustando a la concentración
equivalente de 0,5 McFarland usando un dispositivo DensiCHECK
Plus. Dos microlitros de la concentración recién preparada fueron
depositados como gota en la superficie del agar MRS. Las placas se
mantuvieron a temperatura ambiente durante 30 minutos para
que se absorbieran las gotas y se incubaron en microaerofilia, a
37°C, por 24 h para el desarrollo de las bacterias lácticas.
En paralelo, las cepas patógenas fueron sembradas en agar
nutritivo e incubadas en aerobiosis, a 37ºC, durante 24 h. Se
traspasaron a caldo BHI (Oxoid) y se incubaron en aerobiosis, a
37ºC, por 24 h. Se centrifugaron y el pellet fue resuspendido en
buffer y ajustado a una concentración equivalente de 0.5
McFarland. Se tomó 1 ml de esta suspensión y se mezcló con 9 ml
de agar BHI semisólido fundido. La mezcla se vertió sobre cada placa de agar MRS con las cepas de bacterias lácticas desarrolladas.
Las placas se incubaron en aerobiosis, a 37°C, durante 24 h. Se
midieron los diámetros de los halos de inhibición producidos
alrededor del desarrollo de las bacterias lácticas (originado desde
la gota absorbida).
Cada cepa de bacterias lácticas de la combinación se ajustó por
separado a una concentración de 0,5 McFarland como se describió anteriormente, y se mezclaron en volúmenes iguales. Se tomaron
inóculos de 2 μl de esta mezcla y se sembraron sobre agar MRS.
Las placas fueron incubadas a 37°C, en microaerofilia, durante 24
h. Las cepas patógenas fueron ajustadas a una concentración de 0,5
McFarland, de donde se tomó un volumen de 1 ml y se agregó a un
tubo con 9 ml de agar BHI semisólido fundido, se vertió sobre el
cultivo de cepas de bacterias lácticas ya desarrolladas y se incubó en aerobiosis a 37°C, durante 24 h. Se midió el diámetro de los
halos de inhibición producidos.
Cada cepa de bacteria láctica se ajustó a una concentración de 0,5
McFarland, una cepa fue sembrada sobre agar MRS y se incubó en
microaerofilia, a 37°C, durante 24 h. Se tomó 1 ml de la otra cepa y
se mezcló con 9 ml de agar MRS semisólido fundido, luego se vertió esta mezcla sobre las placas de agar MRS con la primera cepa ya
desarrollada y se incubó en microaerofilia, a 37°C, durante 24 h. El
mismo procedimiento se repitió con las dos cepas en roles
invertidos. Se midió el diámetro de los halos de inhibición
producidos.
Los halos de inhibición producidos fueron clasificados en 4
categorías: sin inhibición, halos de diámetro menor a 0,7 cm;
inhibición leve para diámetros de inhibición entre 0,7 y 1,5 cm;
inhibición moderada para diámetros de inhibición entre 1,6 y 2,5
cm; inhibición fuerte para diámetros de inhibición mayores a 2,5
cm.
Los experimentos de inhibición de patógenos se realizaron en
triplicado y los resultados se expresan como el valor de la media± la desviación estándar (DE)(37,38). Se realizó una Prueba de Kruskal-
Wallis para evaluar diferencia entre medias de los grupos(39).
Valores de p < 0.05 se consideraron estadísticamente
significativos(40). El análisis estadístico fue realizado con el
programa estadístico InfoStat.
Al evaluar la actividad antimicrobiana de las 7 cepas de bacterias
lácticas individuales, se observó que todas presentaron actividad
inhibitoria sobre las cepas patógenas evaluadas. La formación de
halos de inhibición alrededor de las colonias de bacterias lácticas
en forma de gota se puede observar en la Figura 1.

Figura 1: Ejemplo del efecto inhibitorio de la cepa TUCO-L2 sobre la cepa patógena de E. coli enterohemorrágica (EHEC) mediante técnica spot soft agar
Los diámetros de los halos de inhibición producidos por las cepas
de bacterias lácticas se muestran en la Tabla 1. Las cepas de
bacterias lácticas con mayores halos de inhibición sobre E. coli ATCC 25922 fueron TUCO-L2, TUCO-3 y TUCO-5, con un efecto
inhibitorio similar al de la cepa probiótica de referencia L. casei
Shirota. Las cepas TUCO-L2, TUCO-16, TUCO-3 y L. casei Shirota mostraron el mayor efecto inhibitorio sobre EHEC mientras que
las bacterias lácticas con mayores halos de inhibición sobre S.
enterica ATCC 13076 fueron TUCO-L2, TUCO-5E, TUCO-5, TUCO-3
y L. casei Shirota (Tabla 1).
Tabla 1: Efecto inhibitorio de cepas de bacterias lácticas sobre cepas patógenas
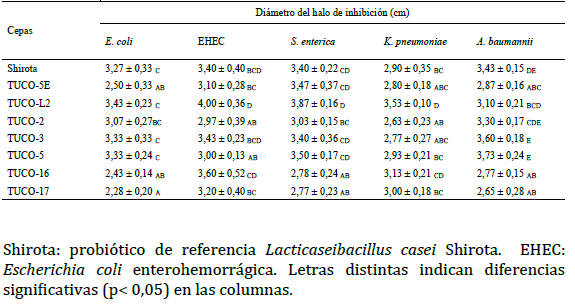
Las cepas TUCO-L2 y TUCO-16 presentaron el mayor efecto
inhibitorio sobre K. pneumoniae ATCC 13883. La cepa TUCO-L2
presentó halos de inhibición significativamente mayores (p < 0,05)
que los observados para L. casei Shirota (Tabla 1). Por otro lado, el
mayor efecto inhibitorio sobre A. baumannii ATCC 19606 fue
producido por las cepas TUCO-5, TUCO-3, TUCO-2 y L. casei Shirota
(Tabla 1).
Los resultados del efecto inhibitorio de las combinaciones de las
cepas de bacterias lácticas sobre las cepas patógenas se muestran
en la Tabla 2. El mayor efecto inhibitorio sobre E. coli ATCC 25922
y sobre EHEC fue producido por la combinación de las cepas
TUCO-5E + TUCO-L2 (Tabla 2). El mayor efecto inhibitorio sobre S.
enterica ATCC 13076 se observó con las combinaciones de cepas
TUCO-5E + TUCO-L2 y TUCO-3 + TUCO-16 (Tabla 2).
Tabla 2: Efecto inhibitorio de las combinaciones de cepas de bacterias lácticas sobre las cepas patógenas
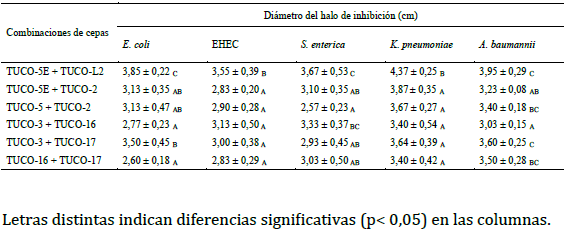
El mayor efecto inhibitorio sobre K. pneumoniae ATCC 13883 fue
producido por la combinación TUCO-5E + TUCO-L2, la cual
presentó un efecto sinérgico en la inhibición de esta cepa
patógena, con halos de inhibición significativamente más grandes
que los producidos por cada cepa de forma individual (p < 0.05).
Las combinaciones TUCO-5E + TUCO-2, TUCO-5 + TUCO-2, y TUCO-
3 + TUCO-17 también tuvieron efectos sinérgicos en la inhibición
de K. pneumoniae, aunque no son tan eficaces como la combinación
TUCO-5E + TUCO-L2 (tabla 2). El mayor efecto inhibitorio sobre la
cepa de A. baumannii ATCC 19606 se observó con las
combinaciones de cepas TUCO-5E + TUCO-L2, TUCO-3 + TUCO-17,
y TUCO-16 + TUCO-17 (Tabla 2). De estas combinaciones, TUCO-5E
+ TUCO-L2 y TUCO-16 + TUCO-17 presentaron efectos sinérgicos
en la inhibición A. baumannii.
Cabe destacar que la combinación TUCO-5E + TUCO-L2 fue la más
efectiva para inhibir el desarrollo de todos los patógenos
evaluados, observándose este efecto en forma más contundente
para K. pneumoniae y A. baumannii.
No se observó efecto inhibitorio (halo de inhibición menor a 0,7 cm de diámetro) entre las cepas TUCO-5E y TUCO-2, ni entre las cepas TUCO-5E y TUCO-L2 (datos no mostrados). Se encontró inhibición leve (halos de inhibición entre 0,7 y 1,5 cm de diámetro) entre las cepas TUCO-2 y TUCO-5, entre las cepas TUCO-3 y TUCO- 16, y entre las cepas TUCO-3 y TUCO-17. Hubo inhibición moderada entre las cepas TUCO-16 y TUCO-17 (halos de inhibición entre 1,6 y 2,5 cm de diámetro). No se encontró inhibición fuerte (halos mayores a 2,5 cm de diámetro) entre las cepas evaluadas.
Los beneficios para la salud proporcionados por los probióticos
son diversos e incluyen su capacidad para incrementar la
resistencia a patógenos(34). En este estudio, se utilizaron cepas de
bacterias lácticas previamente aisladas de la leche de diferentes
animales para evaluar el efecto inhibitorio in vitro de las cepas
individuales, así como de combinaciones contra patógenos
importantes en medicina humana y veterinaria. Se evaluó el efecto
antimicrobiano de las cepas de bacterias lácticas sobre patógenos
gastrointestinales y extraintestinales incluidos en la lista de la
Organización Mundial de la Salud (OMS) de bacterias prioritarias
por su alta resistencia a los antibióticos. Dicha lista incluye
patógenos prioritarios en los que se recomienda centrar la
investigación y el desarrollo de nuevas alternativas
antimicrobianas(41).
Las cepas de bacterias lácticas analizadas tuvieron distintos grados
de inhibición frente a los patógenos evaluados: EHEC, S. enterica, K.pneumoniae, y A. baumannii. La inhibición del desarrollo de los
patógenos en la prueba spot soft agar indica que las cepas de
bacterias lácticas evaluadas producen compuestos activos que
difunden al medio de cultivo y tienen actividad antimicrobiana(42).
Se ha descrito que entre los compuestos con actividad
antimicrobiana producidos por las bacterias lácticas se encuentran
los ácidos orgánicos, peróxido de hidrogeno, óxido nítrico y
bacteriocinas(1,4,12). La naturaleza de los antimicrobianos
producidos por las bacterias de esta investigación todavía no ha
sido dilucidada. Además, fue posible encontrar combinaciones de
cepas de bacterias lácticas con efecto sinérgico para la inhibición in
vitro de diferentes cepas patógenas. Dado que existen pocos
estudios publicados que comparan los efectos de las
combinaciones de cepas de bacterias lácticas potencialmente
probióticas con los efectos de las mismas cepas individualmente,
nuestro trabajo hace un aporte original en este sentido. Se ha
reportado que algunas combinaciones de probióticos tienen mejor
adherencia a la mucosa intestinal que sus cepas individualmente(30), o mayor inhibición de la adherencia de
patógenos a mucosa intestinal humana(31) o porcina(32). Los
resultados presentados acá demuestran además, que
combinaciones apropiadas de cepas de bacterias lácticas pueden
también potenciar el efecto inhibitorio contra patógenos.
Todas las combinaciones de bacterias lácticas tuvieron una
interacción aditiva entre sus cepas constituyentes con respecto a la
inhibición de patógenos entéricos. Notablemente, se observó que
cinco de las combinaciones evaluadas tuvieron efectos sinérgicos
en la inhibición de patógenos extraintestinales: a) se observó efecto sinérgico entre las cepas TUCO-5E + TUCO-L2 para la
inhibición de K. pneumoniae y A. baumannii, b) efecto sinérgico
para TUCO-5E + TUCO-2, TUCO-5 + TUCO-2, y TUCO-3 + TUCO-17
sobre K. pneumoniae y, c) efecto sinérgico para TUCO-16 + TUCO-
17 sobre A. baumannii. Los resultados muestran que la
potenciación del efecto inhibitorio entre cepas combinadas varía
según el patógeno contrastado y, por lo tanto, los productos
probióticos deben diseñarse considerando cada patógeno en
particular. Nuestros resultados están en línea con trabajos previos.
Chapman et al.(37) evaluaron el efecto inhibitorio sobre patógenos
bacterianos de combinaciones de 4 a 15 cepas de bacterias lácticas
empleando el método spot soft agar. Sus resultados mostraron que
solo algunas combinaciones de bacterias lácticas tuvieron mejores
resultados que las cepas individuales, y el efecto inhibitorio fue
específico para cada patógeno. En otro estudio, no se encontraron
diferencias significativas entre las combinaciones y las cepas
individuales para la inhibición de patógenos(43).
E. coli enterohemorrágica (EHEC) es un patógeno de transmisión
alimentaria que puede causar diarrea severa en el hospedador y
complicaciones extraintestinales potencialmente letales, como
falla renal aguda y anormalidades en el sistema nervioso
central(44,45). Es una infección zoonótica, cuyo principal reservorio
son los animales rumiantes, los que suelen ser portadores
asintomáticos(45). En el tratamiento, generalmente se evita el uso
de antibióticos, ya que puede inducir la liberación de toxinas por el
patógeno, en este caso, los probióticos representan una potencial
opción terapéutica, aunque se requieren estudios adicionales(44).
S. enterica no tifoidea es una de las principales causas de
enfermedades transmitidas por alimentos en todo el mundo(46).Distintos serotipos pueden causar infección en el ser humano,
entre ellos, S. enterica serovar Enteritidis tiene la mayor incidencia
y puede infectar tanto a personas como a animales(47). Su principal
fuente de transmisión son las aves y el consumo de sus productos
mal cocinados, la enfermedad en el ser humano se manifiesta
generalmente como una gastroenteritis autolimitante(46,47). Aunque
no es común, se han encontrado cepas de S. enterica con
resistencia a distintos antimicrobianos(46,47).
Entre las cepas de bacterias acido lácticas utilizadas en este
estudio, las cepas L. brevis TUCO-5E(20) y L. salivarius TUCO-L2(23) han demostrado proteger contra la infección de S. enterica Typhimurium en un modelo murino, además, ensayos in vitro
muestran que TUCO-5E tiene la capacidad de excluir y desplazar
patógenos como E. coli y Salmonella del epitelio intestinal(20), y
TUCO-L2 tiene actividad inmunomoduladora(23). La combinación
TUCO-5E + TUCO-L2 fue la más efectiva para inhibir el desarrollo
de todos los patógenos evaluados, además, presentó sinergia en la
inhibición de K. pneumoniae ATCC 13883, y A. baumannii ATCC
19606. Por otro lado, las cepas TUCO-5E y TUCO-L2 no
presentaron inhibición entre ellas. Esto hace que la combinación
TUCO-5E + TUCO-L2 sea un excelente candidato probiótico
multicepa para ser evaluado in vivo en la protección contra la
infección de los distintos patógenos evaluados en este estudio.
K. pneumoniae y A. baumannii son importantes patógenos
nosocomiales que pueden causar diversas infecciones
oportunistas, tanto en humanos como en animales, por ejemplo,
infecciones del tracto respiratorio, infecciones del tracto urinario,
infecciones de tejidos blandos, septicemia y meningitis(48-51). Ambas
especies han desarrollado cepas multirresistentes a los
antibióticos que se propagan rápidamente, esto sumado a su
capacidad de formar biopelículas, hace que sean patógenos
difíciles de controlar y que representen una seria amenaza para la
salud pública(48-50). Por esto, la OMS los clasifica entre las especies
bacterianas patógenas con mayor prioridad en la resistencia a los
antibióticos(41). Las combinaciones TUCO-5E + TUCO-2, TUCO-5 +
TUCO-2, y TUCO-3 + TUCO-17, presentaron un efecto sinérgico en
la inhibición de K. pneumoniae ATCC 13883; mientras que la
combinación TUCO-16 + TUCO-17 presentó un efecto sinérgico en
la inhibición de A. baumannii ATCC 19606. Esto hace que las cuatro
combinaciones sean interesantes candidatos probióticos multicepa para ser evaluado in vivo en la protección contra infecciones
respiratorias y urinarias causadas por patógenos
multirresistentes.
Existen varios métodos in vitro para evaluar el efecto inhibitorio
sobre patógenos de las cepas probióticas(52). Cada técnica puede
dar resultados diferentes, por ejemplo, la cepa con mayor eficacia
inhibitoria puede variar según el método utilizado(34,53). Se ha
descrito, además, diferencias en la misma técnica, por ejemplo, con
la prueba spot soft agar, diferentes investigadores utilizan
distintos medios de cultivo, volúmenes, concentraciones de células
probióticas y patógenas, e incluso distinto modo de medir la zona
de inhibición (diámetro, radio o zona de inhibición de la cepa
probiótica). Por ello, sería de gran importancia confirmar el efecto
sinérgico de las combinaciones, realizando variaciones en
concentraciones de células, medios de cultivos, así como mediante
el empleo de otras técnicas. Por otro lado, es necesario considerar
que las evaluaciones in vitro no repliquen por completo las
condiciones reales in situ de las superficies mucosas como las del
tracto intestinal o respiratorio. Sin embargo, estas técnicas son
herramientas útiles para la selección rápida de cepas con potencial
probiótico, ya que permiten una enorme simplificación del sistema
en estudio ayudando a evaluar un gran número de cepas en busca
de una propiedad probiótica específica. Esto permite seleccionar
las cepas o combinaciones más efectivas para continuar las
investigaciones in vivo, que consumen más tiempo y recursos
económicos(38).
Se encontraron cinco combinaciones con un efecto sinérgico para la inhibición del desarrollo de las cepas patógenas. El efecto sinérgico fue específico para la cepa patógena y se presentó principalmente sobre patógenos del tracto respiratorio y urinario. Las combinaciones que mostraron sinergia son buenas candidatas para el desarrollo de productos probióticos multicepa para la prevención y el tratamiento de infecciones bacterianas específicas. Sin embargo, se necesitan más estudios en aspectos tecnológicos y de formulación y estudios in vivo para demostrar su eficacia y seguridad en los hospedadores en los que se pretenda su uso.
Esta investigación fue financiada por el proyecto FONDEF ID20I10114.
1. Ouwehand AC, Forssten S, Hibberd AA, Lyra A, Stahl B. Probiotic approach to prevent antibiotic resistance. Annals of Medicine. 2016; 48(4):246–255. Doi:10.3109/07853890.2016.1161232
2. Ghosh C, Sarkar P, Issa R, Haldar J. Alternatives to conventional antibiotics in the era of antimicrobial resistance. Trends Microbiol. 2019; 27(4):323 – 327. Doi:10.1016/j.tim.2018.12.010
3. Food and Agriculture Organization & World Healt Organization (FAO / WHO). Probiotics in food: Health and nutricional properties and guidelines for evaluation. FAO Food and Nutritional Paper 85 [Internet] 2006 [Consultado 15 Ago 2022] Disponible en: http://www.fao.org/3/a0512e/a0512e.pdf
4. Hill C, Guarner F, Reid G, Gibson GR, Merenstein DJ, Pot B, Morelli L, Canani RB, Flint HJ, Salminen S, Calder PC, Sanders ME. Expert consensus document: the international scientific association for probiotics and prebiotics consensus statement on the scope and appropriate use of the term probiotic. Nat Rev Gastroenterol Hepatol. 2014: 11:506-514. Doi:10.1038/nrgastro.2014.66
5. Reid G, Gadir AA, Dhir R. Probiotics: reiterating what they are and what they are not. Front Microbiol. 2019; 10:424. Doi:10.3389/fmicb.2019.00424
6. Binda S, Hill C, Johansen E, Obis D, Pot B, Sanders ME, et al. Criteria to qualify microorganisms as “Probiotic” in foods and dietary supplements. Front Microbiol. 2020; 11. Doi:10.3389/fmicb.2020.01662
7. Forssten SD, Laitila A, Maukonen J, Ouwehand AC. Probiotic triangle of success; strain production, clinical studies and product development. FEMS Microbiol Lett. 2020; 367(19). Doi:10.1093/femsle/fnaa167
8. Fontana L, Bermudez-Brito M, Plaza-Diaz J, Muñoz-Quezada S, Gil A. Sources, isolation, characterisation and evaluation of probiotics. B J Nutr. 2013; 109(S2): S35–S50. Doi:10.1017/S0007114512004011
9. Jugan MC, Rudinsky AJ, Parker VJ, Gilor C. Use of probiotics in small animal veterinary medicine. J Am Vet Med. 2017; 250(5): 519–528. Doi:10.2460/javma.250.5.519
10. King S, Tancredi D, Lenoir-Wijnkoop I, Gould K, Vann H, Connors G, et al. Does probiotic consumption reduce antibiotic utilization for common acute infections? A systematic review and meta-analysis. Eur J Public Health. 2019; 29(3): 494-499. Doi:10.1093/eurpub/cky185
11. Markowiak P, Slizewska K. Effects of probiotics, prebiotics, and synbiotics on human health. Nutrients. 2017; 9(9). Doi:10.3390/nu9091021
12. Mathipa MG, Thantsha MS. Probiotic engineering: towards development of robust probiotic strains with enhanced functional properties and for targeted control of enteric pathogens. Gut pathog. 2017; 9: 28. Doi:10.1186/s13099-017-0178-9
13. McFarland LV, Huang Y, Wang L, Malfertheiner P. Systematic review and meta-analysis: multi-strain probiotics as adjunct therapy for Helicobacter pylori eradication and prevention of adverse events. United Eur Gastroenterol J. 2016; 4(4): 546–561. Doi:10.1177/2050640615617358
14. Sornplang P, Piyadeatsoontorn S. Probiotic isolates from unconventional sources: a review. J Anim Sci Technol. 2016; 58(1). Doi:10.1186/s40781- 016-0108-2
15. Zielińska D, Kolożyn-Krajewska D. Food-origin lactic acid bacteria may exhibit probiotic properties: review. BioMed Res Int. 2018. Doi:10.1155/2018/5063185
16. Fernández L, Langa S, Martín V, Maldonado A, Jiménez E, Martín R, et al. The human milk microbiota: Origin and potential roles in health and disease. Pharmacol Res. 2013; 69(1): 1–10. Doi:10.1016/j.phrs.2012.09.001
17. Makete G, Aiyegoro OA, Thantsha MS. Isolation, identification and screening of potential probiotic bacteria in milk from South African Saanen goats. Probiotics Antimicrob Proteins. 2016; 9(3): 246–254. Doi:10.1007/s12602-016-9247-5
18. Reuben RC, Roy PC, Sarkar SL, Rubayet Ul Alam A, Jahid IK. Characterization and evaluation of lactic acid bacteria from indigenous raw milk for potential probiotic properties. J dairy sci. 2020; 103(2): 1223–1237. Doi: 10.3168/jds.2019-17092
19. Martín R, Delgado S, Maldonado A, Jiménez E, Olivares M, Fernández L, et al. Isolation of lactobacilli from sow milk and evaluation of their probiotic potential. J Dairy Res. 2009; 76: 418–425. Doi:10.1017/S0022029909990124
20. Quilodrán-Vega SR, Villena J, Valdebenito J, Salas MJ, Parra C, Ruiz A, et al. Isolation of lactic acid bacteria from swine milk and characterization of potential probiotic strains with antagonistic effects against swineassociated gastrointestinal pathogens. Can J Microbiol. 2016; 62(6): 514–524. Doi:10.1139/cjm-2015-0811
21. Martín R, Olivares M, Pérez M, Xaus J, Torre C, Fernández L, et al. Identification and evaluation of the probiotic potential of lactobacilli isolated from canine milk. Vet J. 2010; 185(2): 193–198. Doi:10.1016/j.tvjl.2009.04.014
22. Fernandez L, Martínez R, Cruz M, Arroyo R, Rodríguez JM. Characterization of Lactobacillus rhamnosus MP01 and Lactobacillus plantarum MP02 and assessment of their potential for the prevention of gastrointestinal infections in an experimental canine model. Front Microbiol. 2019; 10. Doi:10.3389/fmicb.2019.01117
23. Quilodrán-Vega S, Albarracin L, Mansilla M, Arce L, Zhou B, Islam MA, et al. Functional and genomic characterization of Ligilactobacillus salivarius TUCO-L2 isolated from Lama glama milk: a promising immunobiotic strain to combat infections. Front Microbiol. 2020; 11: 608752. Doi:10.3389/fmicb.2020.608752
24. Forssten SD, Ouwehand AC. Simulating colonic survival of probiotics in single-strain products compared to multi-strain products. Microb Ecol Health Dis. 2017; 28(1). Doi:10.1080/16512235.2017.1378061
25. Ouwehand AC, Invernici MM, Furlaneto FA, Messora MR. Effectiveness of multi-strain versus single-strain probiotics: current status and recommendations for the future. J Clin Gastroenterol. 2018; 52(S1): S35– S40. doi:10.1097/mcg.0000000000001052
26. Timmerman HM, Koning CJ, Mulder L, Rombouts FM, Beynen AC. Monostrain, multistrain and multispecies probiotics - A comparison of functionality and efficacy. Int J Food Microbiol. 2004; 96(3): 219 - 233. Doi:10.1016/j.ijfoodmicro.2004.05.012
27. Chapman CM, Gibson GR, Rowland I. Health benefits of probiotics: are mixtures more effective than single strains? Eur J Nutr. 2011; 50(1): 1– 17. Doi:10.1007/s00394-010-0166-z
28. Gómez-Gallego C, Junnila J, Männikkö S, Hämeenoja P, Valtonen E, Salminen S, et al. A canine-specific probiotic product in treating acute or intermittent diarrhea in dogs: a double-blind placebo-controlled efficacy study. Vet Microbiol. 2016; 197: 122–128. Doi:10.1016/j.vetmic.2016.11.015
29. McFarland LV, Ship N, Auclair J, Millette M. Primary prevention of Clostridium difficile infections with a specific probiotic combining Lactobacillus acidophilus, L. casei, and L. rhamnosus strains: assessing the evidence. J Hosp Infect. 2018; 99(4): 443 – 452 Doi:10.1016/j.jhin.2018.04.017.
30. Collado MC, Meriluoto J, Salminen S. Development of new probiotics by strain combinations: is it possible to improve the adhesion to intestinal mucus?, J Dairy Sci. 2007; 90(6): 2710–2716. Doi:10.3168/jds.2006-456
31. Collado MC, Meriluoto J, Salminen S. In vitro analysis of probiotic strain combinations to inhibit pathogen adhesion to human intestinal mucus. Food Res Int. 2007; 40(5): 629 – 636. Doi:10.1016/j.foodres.2006.11.007
32. Collado MC, Grześkowiak Ł, Salminen S. Probiotic strains and their combination inhibit in vitro adhesion of pathogens to pig intestinal mucosa. Curr Microbiol. 2007; 55(3): 260 – 265. Doi:10.1007/s00284- 007-0144-8
33. Scalfaro C, Iacobino A, Nardis C, Franciosa G. Galleria mellonella as an in vivo model for assessing the protective activity of probiotics against gastrointestinal bacterial pathogens. FEMS Microbiol Lett. 2017; 364(7): 1 – 6. Doi:10.1093/femsle/fnx064, 46 - 42
34. Fijan S, Šulc D, Steyer A. Study of the in vitro antagonistic activity of various single-strain and multi-strain probiotics against Escherichia coli. Int J Environ Res Public Health. 2018; 15(7). doi:10.3390/ijerph15071539
35. Altarugio R, Vellano IH, Moraes AC, Milbradt EL, Andreatti-Filho RL, Guimarães-Okamoto PT, et al. In vitro probiotic selection and characterization of Lactobacillus spp. isolated from healthy domesticated Turkeys. J Appl Poult Res. 2017; 27(1): 81–91. doi:10.3382/japr/pfx045
36. Jimenez-Trigos E, Toquet M, Barba M, Gómez-Martín Á, Quereda JJ, Bataller E. Search of antimicrobial lactic acid bacteria from Salmonellanegative dogs. BMC Vet Res. 2022;18(1): 1 – 12. doi:10.1186/s12917- 021-03070-x
37. Chapman CM, Gibson GR., Rowland I. In vitro evaluation of single- and multi-strain probiotics: inter-species inhibition between probiotic strains, and inhibition of pathogens. Anaerobe. 2012; 18(4): 405 – 413. Doi:10.1016/j.anaerobe.2012.05.004
38. Shokryazdan P, Sieo CC, Kalavathy R, Liang JB, Alitheen NB, Faseleh Jahromi M, et al. Probiotic potential of Lactobacillus strains with antimicrobial activity against some human pathogenic strains. BioMed Res Int. 2014; 927268. doi: 10.1155/2014/927268
39. Amstrong RA, Hilton AC. Statistical analysis in microbiology: Statnotes. USA: Wiley-Blackwell; 2011.
40. Lorowitz W, Saxton E, Sondossi M, Nakaoka K. Integrating statistics with a microbiology laboratory activity. Microbiology education. 2005; 6: 14– 19.
41. World Healt Organization (WHO). Global priority list of antibioticresistant bacteria to guide research, discovery, and development of new antibiotics. Geneva, Switzerland [Imternet] 2017 [Consultado 20 Mar 2020] Disponible en: https://www.who.int/medicines/publications/WHO-PPLShort_ Summary_25Feb-ET_NM_WHO.pdf
42. Tejero-Sariñena S, Barlow J, Costabile A, Gibson GR, Rowland I. In vitro evaluation of the antimicrobial activity of a range of probiotics against pathogens: evidence for the effects of organic acids. Anaerobe. 2012; 18(5): 530-538. doi: 10.1016/j.anaerobe.2012.08.004
43. Chapman CM, Gibson GR, Todd S, Rowland I. Comparative in vitro inhibition of urinary tract pathogens by single- and multi-strain probiotics. Eur J Nutr. 2013; 52(6): 1669–1677. Doi:10.1007/s00394- 013-0501-2
44. Hwang SB, Chelliah R, Kang JE, Rubab M, Banan-MwineDaliri E, Elahi F, Oh DH. Role of recent therapeutic applications and the infection strategies of Shiga toxin-producing Escherichia coli. Front Cell Infect Microbiol. 2021; 11: 614963. Doi:10.3389/fcimb.2021.614963
45. Kim J-S, Lee M-S, Kim JH. Recent Updates on outbreaks of Shiga Toxin- Producing Escherichia coli and its potential reservoirs. Front Cell Infect Microbiol. 2020; 10:273. doi: 10.3389/fcimb.2020.00273, 56
46. Fernández J, Guerra B, Rodicio MR. Resistance to Carbapenems in Non- Typhoidal Salmonella enterica Serovars from Humans, Animals and Food. Vet Sci. 2018; 5(2): 40. doi:10.3390/vetsci5020040
47. Retamal P, Fresno M, Dougnac C, Gutierrez S, Gornall V, Vidal R, et al. Genetic and phenotypic evidence of the Salmonella enterica serotype Enteritidis human-animal interface in Chile. Front Microbiol. 2015; 6:464. doi: 10.3389/fmicb.2015.00464
48. Yang F, Deng B, Liao W, Wang P, Chen P, Wei J. High rate of multiresistant Klebsiella pneumoniae from human and animal origin Infect Drug Resist. 2019; 12:2729-2737. doi: 10.2147/IDR.S219155.
49. Wang G, Zhao G, Chao X, Xie L, Wang H. The Characteristic of Virulence, Biofilm and Antibiotic Resistance of Klebsiella pneumoniae. Int J Environ Res Public Health. 2020; 17(17):6278. doi:10.3390/ijerph17176278
50. Asif M, Alvi IA, Rehman SU. Insight into Acinetobacter baumannii: pathogenesis, global resistance, mechanisms of resistance, treatment options, and alternative modalities. Infect Drug Resist. 2018; 11: 1249– 1260. Doi: 10.2147/IDR.S166750
51. Van der Kolk JH, Endimiani A, Graubner C, Gerber V, Perreten V. Acinetobacter in Veterinary Medicine with emphasis on A. baumannii. J Glob Antimicrob Resist. 2019;16: 59 – 71.
52. Fijan S. Antimicrobial effect of probiotics against common pathogens. En: Rao V, Rao LG, editors. Probiotics and Prebiotics in Human Nutrition and Health. IntechOpen. 2016. p.191-211. Doi:10.5772/63141
53. Halder D, Mandal M, Chatterjee SS, Pal, NK, Mandal S. Indigenous probiotic Lactobacillus isolates presenting antibiotic like activity against human pathogenic bacteria. Biomedicines. 2017; 5(2). doi: 10.3390/biomedicines5020031
Fecha de recibido: 06/09/2022
Fecha de aceptado para su publicación: 31/10/2022